【迁移】从ENI数据库下载fastq文件
Last updated on March 19, 2024 pm
从 ENI 数据库下载
进入ENA Browser,搜索对应的GSE号,进入study项目,选择TSV格式的Download report。
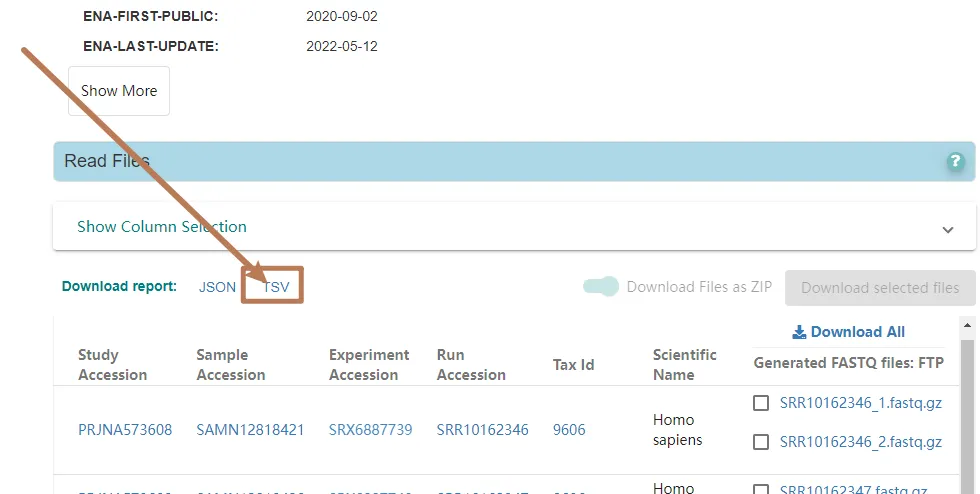
从TSV表格中提取下载链接,一行一个写入url.txt,前面加上ftp://,接着使用wget -c -i url.txt下载
批量重命名脚本:
1 | |
使用 Aspera 下载
Aspera是IBM公司的一款私有专利的高速传输软件,据说能充分利用现有的 WAN 基础设施和通用硬件,传输速度比 FTP 和 HTTP 快达数百倍。
安装aspera
1 | |
获取下载地址
将
ftp://ftp.sra.ebi.ac.uk/vol1/之类的地址的前缀
换成era-fasp@fasp.sra.ebi.ac.uk:/vol1/
进行下载
1 | |
1 | |
- 似乎确实挺快的,可以直连,没有不可名状的干扰
从 NCBI 数据库下载
安装依赖
1 | |
批量下载SRA文件
- mkdir upload/zl_liu/sra/GSE172205
- cd upload/zl_liu/sra/GSE172205
- 通过 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=<GSE_ID> 得到 <SRP_ID>
- 通过 https://www.ncbi.nlm.nih.gov/Traces/study/?acc=<SRP_ID> 搜索 <SRP_ID>
- 下载 Total 的 Accession List,上传到 upload/zl_liu/sra/GSE172205 目录下
- vdb-config -i 设置http代理,网络好也可以不设置
- prefetch --option-file SRR_Acc_List.txt
批量转换为FASTQ文件
1 | |
1 | |
后续分析
【迁移】从ENI数据库下载fastq文件
https://hexo.limour.top/cong-ENI-shu-ju-ku-xia-zai-fastq-wen-jian