2022-09-25-【迁移】从ENI数据库下载fastq文件.md 3.3 KB
title: 【迁移】从ENI数据库下载fastq文件 urlname: cong-ENI-shu-ju-ku-xia-zai-fastq-wen-jian date: 2022-09-25 19:32:17
tags: ['ENI', 'NCBI-GEO', 'fastq', 'SRA']
从 ENI 数据库下载
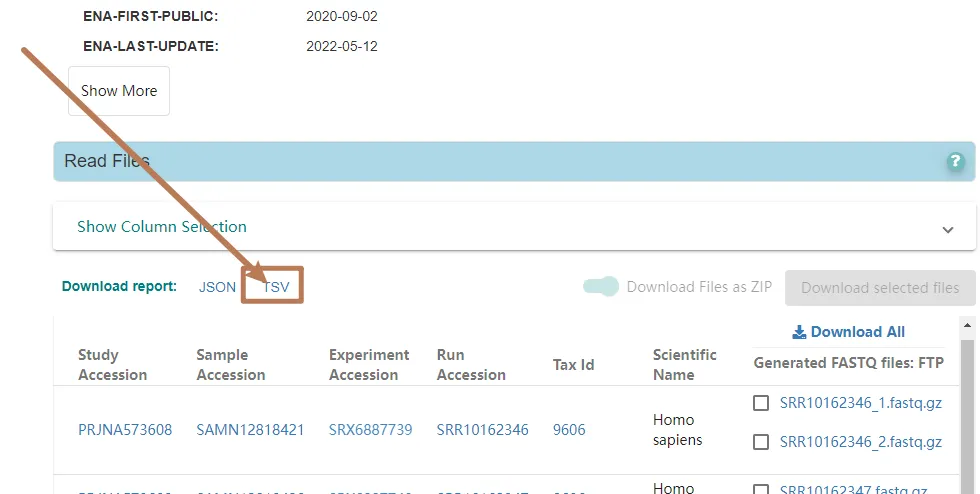
进入ENA Browser,搜索对应的GSE号,进入study项目,选择TSV格式的Download report。
从TSV表格中提取下载链接,一行一个写入url.txt,前面加上ftp://,接着使用wget -c -i url.txt下载
批量重命名脚本:
ls *.fastq.gz | cut -d '_' -f 1 | while read i ;do (echo ${i}_1*.gz' will be moved to '${i}_S1_L001_R1_001.fastq.gz);done
ls *.fastq.gz | cut -d '_' -f 1 | while read i ;do (echo ${i}_2*.gz' will be moved to '${i}_S1_L001_R2_001.fastq.gz);done
ls *.fastq.gz | cut -d '_' -f 1 | while read i ;do (mv ${i}_1*.gz ${i}_S1_L001_R1_001.fastq.gz;mv ${i}_2*.gz ${i}_S1_L001_R2_001.fastq.gz);done
使用 Aspera 下载
Aspera是IBM公司的一款私有专利的高速传输软件,据说能充分利用现有的 WAN 基础设施和通用硬件,传输速度比 FTP 和 HTTP 快达数百倍。
安装aspera
# conda create -n linux -c conda-forge tree
conda activate linux
conda install -c hcc aspera-cli -y
获取下载地址
将
ftp://ftp.sra.ebi.ac.uk/vol1/之类的地址的前缀 换成era-fasp@fasp.sra.ebi.ac.uk:/vol1/
进行下载
nano SRR12303173DL.sh && chmod +x SRR12303173DL.sh
./SRR12303173DL.sh
#!/bin/bash
source activate linux
dsa=$CONDA_PREFIX/etc/asperaweb_id_dsa.openssh
runAscp="ascp -qT -l 80m -P 33001 -i $dsa era-fasp@fasp.sra.ebi.ac.uk:"
#保存路径
savedir=/home/jovyan/upload/rawData/GSM4685043
mkdir -p $savedir
#设置需要下载的文件
## SRR12303173
tempdir=$savedir'/SRR12303173'
mkdir $tempdir
$runAscp'/vol1/fastq/SRR123/073/SRR12303173/SRR12303173_1.fastq.gz' $tempdir'/dl_1.fastq.gz'
$runAscp'/vol1/fastq/SRR123/073/SRR12303173/SRR12303173_2.fastq.gz' $tempdir'/dl_2.fastq.gz'
似乎确实挺快的,可以直连,没有不可名状的干扰
从 NCBI 数据库下载
安装依赖
conda create -n sra_tools -c bioconda sra-tools
conda activate sra_tools
conda install -c conda-forge pigz -y
prefetch
批量下载SRA文件
- mkdir upload/zl_liu/sra/GSE172205
- cd upload/zl_liu/sra/GSE172205
- 通过 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc= 得到
- 通过 https://www.ncbi.nlm.nih.gov/Traces/study/?acc= 搜索
- 下载 Total 的 Accession List,上传到 upload/zl_liu/sra/GSE172205 目录下
- vdb-config -i 设置http代理,网络好也可以不设置
- prefetch --option-file SRR_Acc_List.txt
批量转换为FASTQ文件
#!/bin/sh
#任务名
TASKN=GSE172205
#设置根目录
ROOTDIR=/home/jovyan/upload/zl_liu/sra/GSE172205
#设置CleanData存放目录
CLEAN=$ROOTDIR/$TASKN
mkdir $CLEAN
for file in `cat SRR_Acc_List.txt`
do
echo $file
mkdir $CLEAN/$file
cd $CLEAN/$file
fasterq-dump --split-3 $ROOTDIR/$file -e 16
pigz -p 16 *
done
nano 11.sh
chmod +x 11.sh
./11.sh